2021年5月10日,同济大学附属同济医院/脊柱脊髓损伤再生修复教育部重点实验室程黎明、朱融融教授在国际知名学术期刊Nature communications杂志上在线发表了题为《Deep learning-based predictive identification of neural stem cell differentiation.》的研究论文,并入选该杂志“干细胞”领域编辑推荐文章(Editors’ Highlights in ‘Stem cells’ area),详见https://www.nature.com/collections/pkgztbmgly。

图1. 文章入选Nature Communications杂志Editors’ Highlights in ‘Stem cells’ area
神经干细胞(NSCs)具有自我更新和三系分化的潜能,能被诱导分化成神经元、星形胶质细胞和少突胶质细胞,具有重要的神经中枢神经系统疾病(CNS)再生修复研究和应用价值。将NSCs定向分化为神经元一直是该领域的重要研究方向,常见的诱导药物包括有神经营养因子、小分子药物或激素等。传统的药物筛选鉴定方法如免疫荧光等,存在周期长、成本高、背景干扰等问题,寻找更高效的NSCs分化预测方法对于该领域的研究进展至关重要。
该研究构建了一种基于高通量流式单细胞成像技术的NSCs分化命运预测系统,并验证了其对于不同形式、不同作用机制诱导剂的作用下NSCs命运的预测效果。首先用流式细胞术标记获取不同分化方向的NSCs单细胞图像(图2a),再使用卷积神经网络(CNN)提取图像的特征,形成了一个结构良好的图像分类模型(图2b)。该工作构建的模型具有很高的普适性,包括实验室常用的神经营养因子、激素等,以及具有创新前沿性的纳米颗粒等。且该系统仅仅用明场单细胞图像评估,就能够在24小时的预判中获得很高的准确度(图2c)。
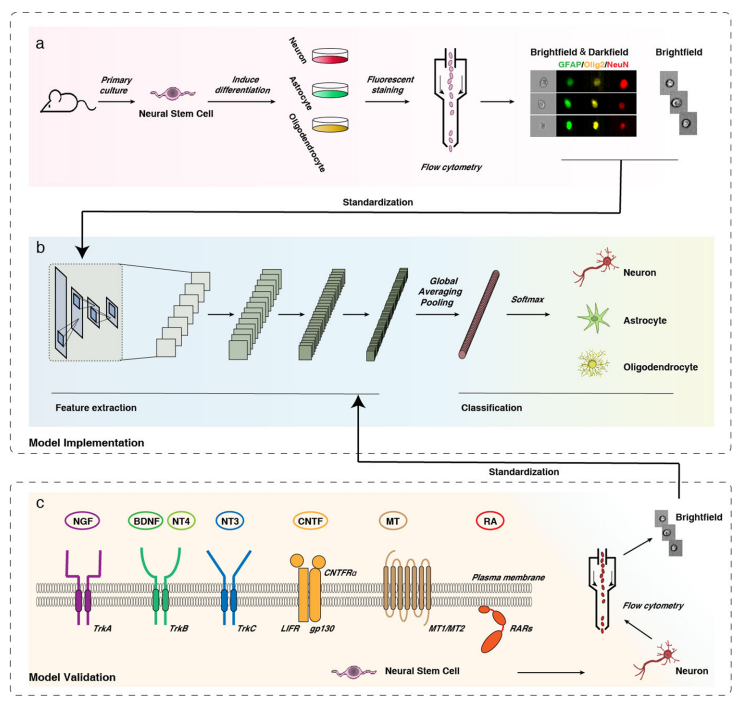
图2. 基于深度学习的NSCs分化方向预测系统的构建示意图
结果表明研究建立的预测系统在NSCs不同分化天数和分化方向均有较高的预测精度,能够利用流式单细胞明场图像精准预测出因子/小分子/纳米材料短时间处理细胞后的未来分化方向,其中,神经元、星形胶质细胞和少突胶质细胞方向最短分别仅需耗时1天、12小时和1天,预测得到分化比例分别为0.827、0.959和0.806,对独立测试集的预测结果显示细胞分化比例与免疫荧光染色实验获得的结果的比例高度一致(图3a-c)。另外,类激活映射技术(CAM)结果表明系统在判断时能够自发将注意力聚焦在细胞,包括边缘和内部细胞器(图3d)。该平台相比传统的分子实验手段和评估方法,具有时间大幅缩短、操作简便、抗干扰、前瞻性和高精度的优势,能够克服现有实验室方法和技术的诸多限制。
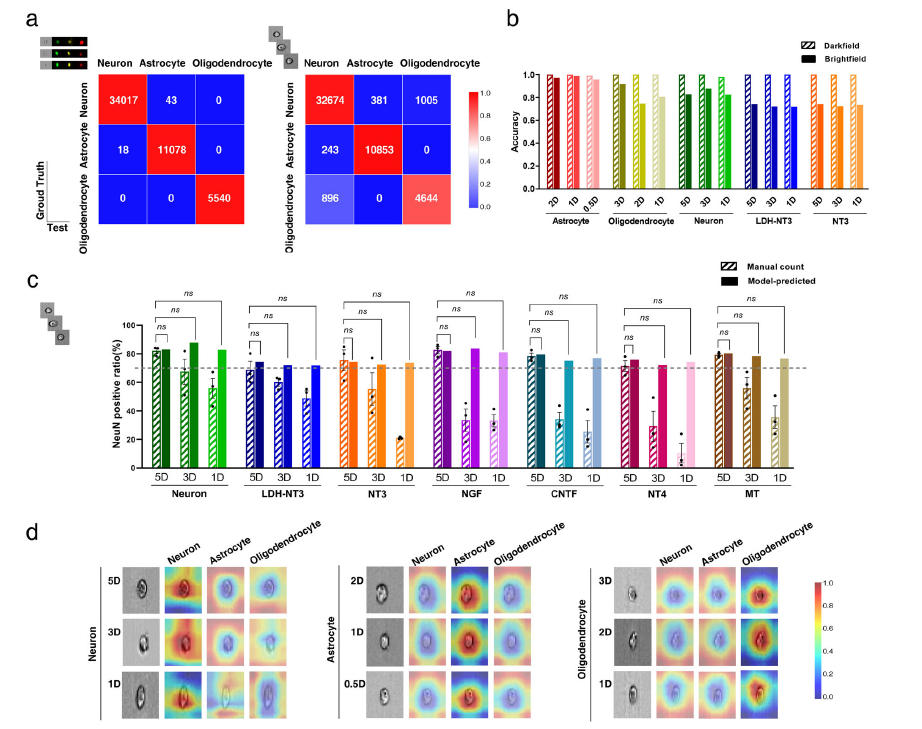
图3. 深度学习对NSCs在不同条件下的分化命运的预测
该研究成果朱颜菁、黄蕊奇为共同第一作者,程黎明、朱融融教授为共同通讯作者。研究提供了一个精度高、误报率低、速度快、适用性广、操作简单、经济实用的NSC分化命运预测平台,从而可以大幅度简化和加快NSCs分化相关新型诱导方式的鉴定,在干细胞治疗CNS的研究方面有极高的应用价值。